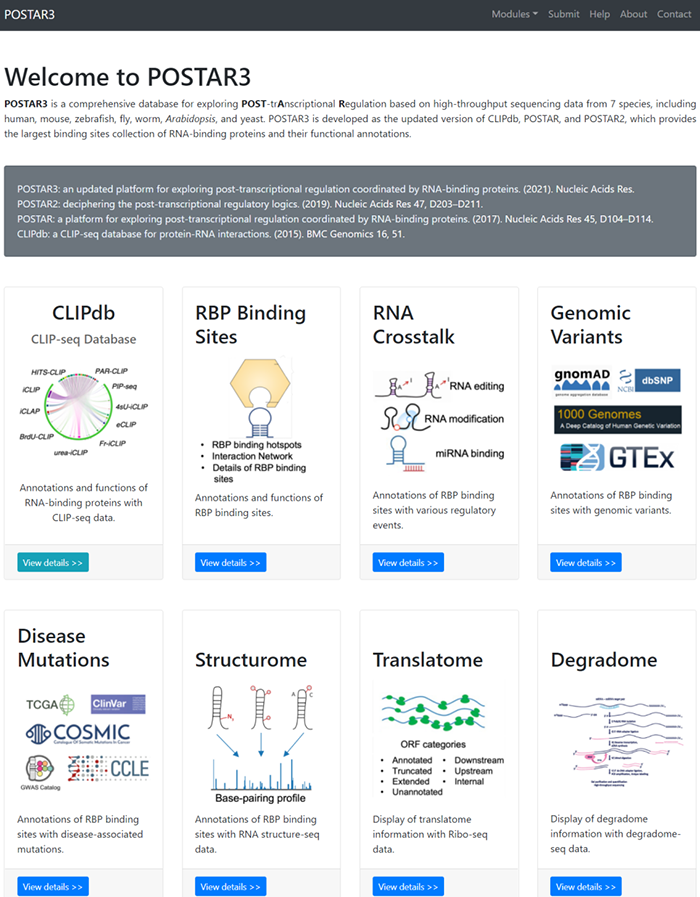
RNA结合蛋白(RBP)在转录后的调节中起着关键的作用。准确识别来自不同种属的多种细胞系和组织类型中的RBP结合位点对于了解RBP在生理和病理条件下的调节机制具有非常重要的作用。
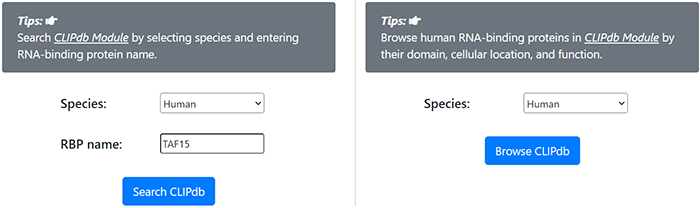
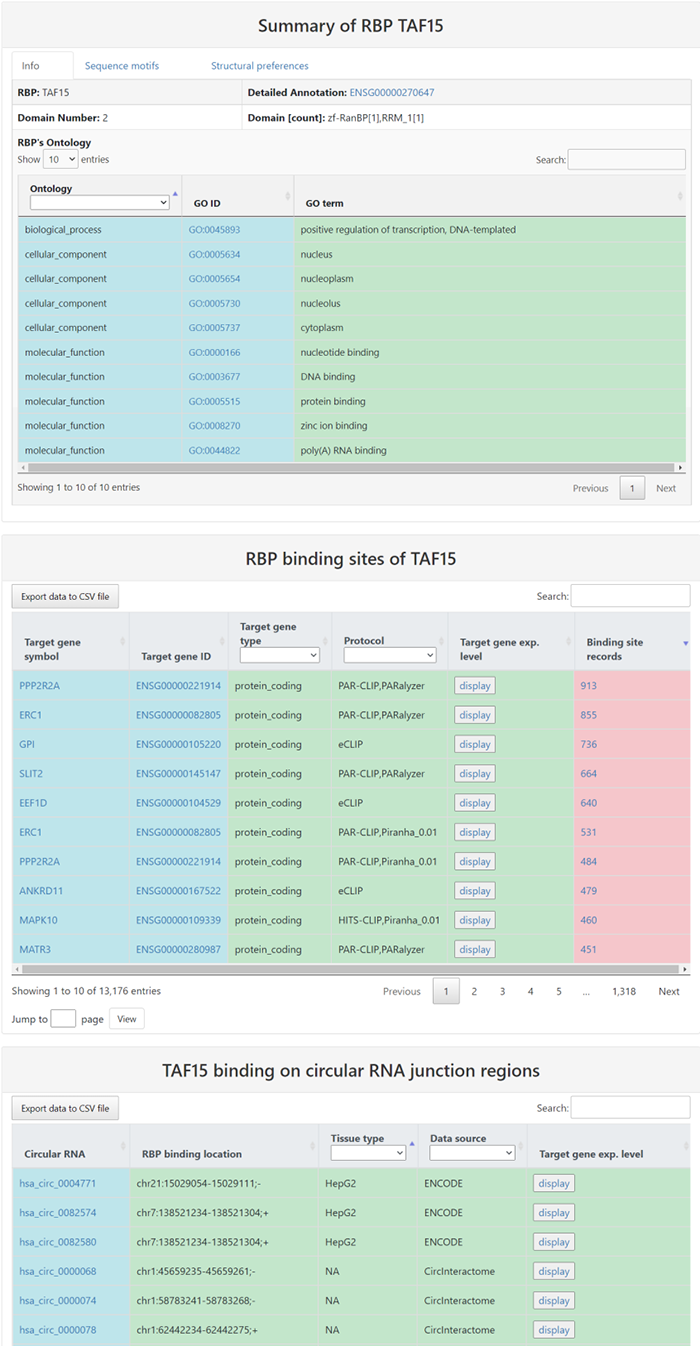
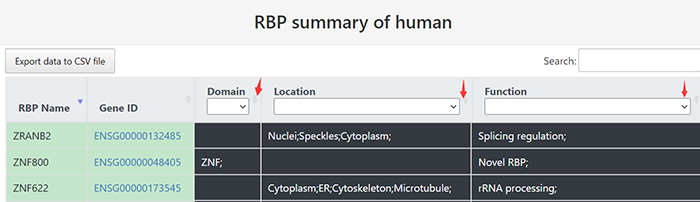
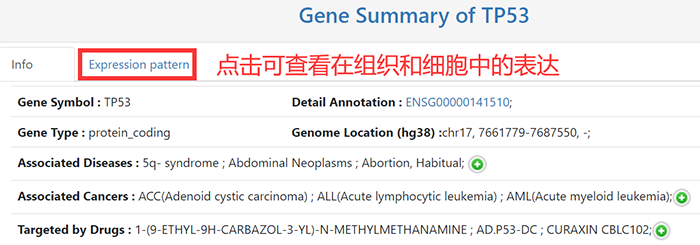
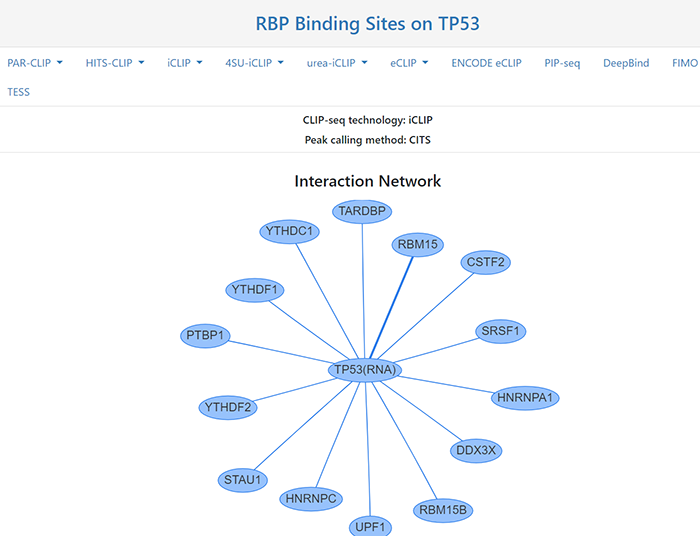
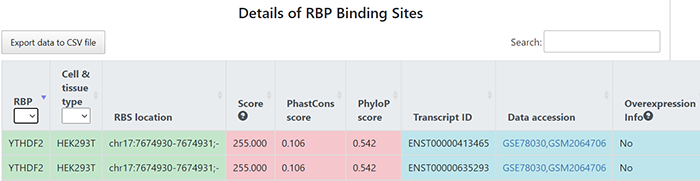
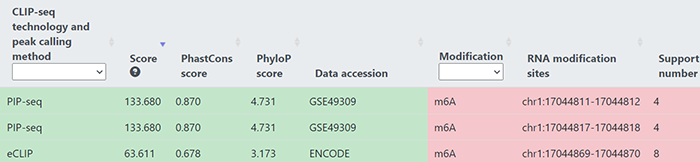
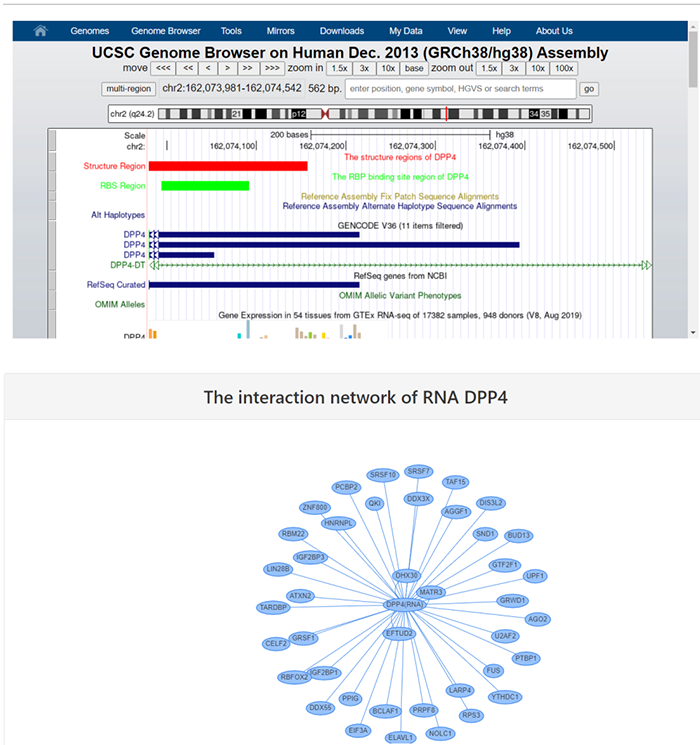
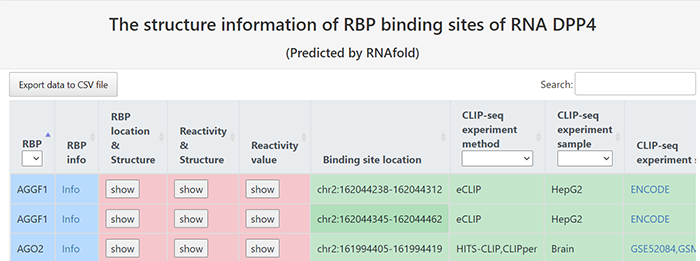
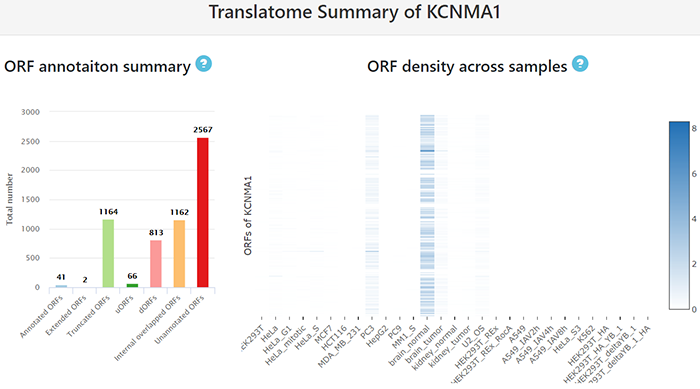
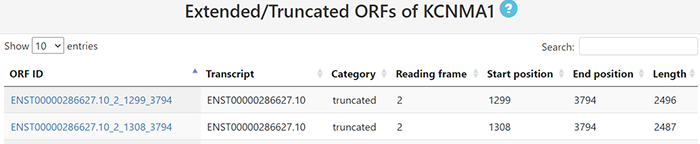
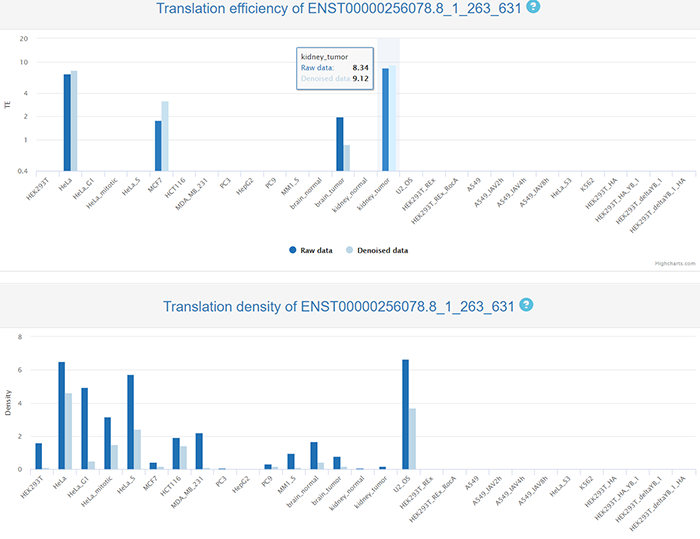
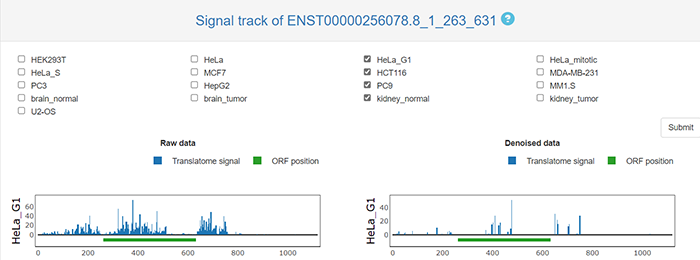
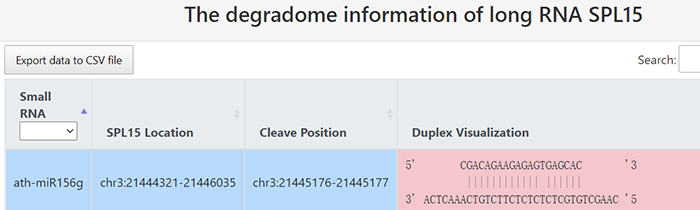
2021年8月17日,清华大学生命科学学院的鲁志教授和复旦大学类脑智能科技研究院的杨禹丞副教授在国际著名期刊《Nucleic Acids Research》上发表了题为《POSTAR3: an updated platform for exploring post-transcriptional regulation coordinated by RNA-binding proteins》的论文,正式发表了他们的POSTAR3数据库。POSTAR3数据库也是之前POSTAR和POSTAR2的升级版本。POSTAR系列数据库利用公开的大规模CLIP-seq数据集和外部功能基因组注释,以生成RBP结合位点的综合图谱及其与其他调节机制以及功能变异之间关联。在此基础上,POSTAR3改进了数据收集、注释和分析的方法,支持多物种转录后调控的注释。POSTAR3数据库的网址为:http://postar.ncrnalab.org/index.html。下面将由本工来对POSTAR3的功能和使用进行介绍。CLIPdb模块可为RNA结合蛋白提供各种注释,包括RNA识别域、Gene Ontology、序列基序和结构偏好。用户可以用来查询RNA结合蛋白的所有结合位点和靶基因的表达水平。用户在左边的栏目可通过选择物种并输入RNA结合蛋白名称来进行搜索,也可在右边的栏目,根据结合域、细胞定位和功能来浏览CLIPdb模块中的人类RNA结合蛋白。在左边的栏目中点击“Search CLIPdb”即可查看关于TAF15蛋白的各种信息,包括结合域数目、Gene Ontology信息、结合的靶基因及其类型和结合位点、研究的方法、靶基因的表达水平(点击后即可跳转到新的页面展示),还有与circRNA的结合情况(包括结合的位置、组织类型、数据来源、靶基因的表达水平)。当然,大家也可点击右侧的栏目以自由浏览,点击下拉栏则可进行筛选,也可在右上角的搜索框中进行搜索。RBP Binding Sites模块利用不同的CLIP-seq技术和相应的peak calling方法,以表格和网络的形式展示靶基因的注释和功能,包括基因组位置、RNA的表达模式以及RNA与RBPs的相互作用。在这个模块大家可以选择物种并输入RNA的symbol以进行搜索。点击搜索后我们可以看到RNA的各种基本信息,包括位置、相关疾病、和靶向该RNA的药物。还可以查看基于不同CLIP方法构建的相互作用网络。以及与不同RBP之间的结合情况,其中Score表示call peak时得到的RBP结合分数,而PhastCons score和Phylop score则表示使用不同软件所得到的RBP结合位点的平均保守分数。RNA Crosstalk模块可提供RBP结合位点周围的RNA转录后调控的相互作用,包括miRNA靶点、RNA修饰和RNA编辑。RBPs还可参与各种转录后调控事件,并在这些过程中发挥重要作用,因此,用户可以在该模块中研究关于这些调控事件中的潜在Crosstalk。在该模块中,用户可以查看经实验验证或者预测到的RBP 结合位点内的miRNA结合情况。Genomic Variants模块将单核苷酸变异和性状相关的突变与RBP结合位点整合到了一起,以帮助研究那些可调节分子机制和不同性状的SNPs。由于RBPs与SNP的识别和功能有关,理解不同变异体和RBP结合位点之间的关联,有助于发现更多的分子机制。这部分的基本使用方法和上面的一致,主要增添了关于参考序列、变异序列、SNP位置和组织类型的信息。大家可以比较变异前后的结合score来判断SNP对RBP结合能力的影响。Disease Mutations模块将突变和RBP结合位点中与疾病相关的SNVs整合在了一起,可帮助大家深入了解潜在的调控机制和人类疾病背后的SNVs。这部分的数据主要有GWAS、ClinVar、TCGA、COSMIC、CCLE、HmtDB。具体使用方法和上述模块基本一致。Structurome模块提供由不同算法所预测的RNA二级结构,同时添加了RBP结合点的二级结构分析数据。结合位点也至少扩展到150nt,并同时提供了从实验中得到的反应性数据和二级结构模型。这个模块为为大家提供了直接浏览查看UCSC中该基因相关信息的便利,还有该基因的相互作用网络。以及该基因RBP结合位点的信息,大家可以可以点击红色方框中的show,以查看RBP的位置和结构、实验中的反应性情况和结构以及具体的反应性数据。Translatome模块使用Ribo-seq的数据来描述翻译的相关情况。输入基因进行搜索后所得到的结果中,左边的直方图表示不同类型的ORF的数量,右边的热图表示不同实验条件下的每个开放阅读框架的翻译情况。展示的表格则包含不同类型ORF的信息,包括分类、阅读框、起始终止位点和长度。点击ORF ID栏中的蓝色id则可跳转查看该ORF的密度(density)、翻译效率、所鉴定到的翻译区域及核糖体分析信号。Degradome模块提供了小RNA和其他类型的RNA之间的结合,这种结合可导致其他靶标RNA发生降解,并通过Degradome-seq数据进行了验证(解螺旋之前介绍starBase数据库的时候也有介绍类似的功能模块)。这部分先是展示了所输入基因的相互作用网络,然后用表格展示了基因在基因组中的位置和裂解发生的位置(Cleave Position),并用在Duplex visualization中用简单的碱基序列图进行了展示。
文章来源:解螺旋